

哺乳动物的基因组中,约40-50%的区域是由DNA重复序列构成的转座元件。部分转座元件具有转录调控活性,在神经发育中发挥重要作用。作为转座元件的一大类,短散在核元件(short interspersed nuclear elements, SINEs)包含锌指蛋白CTCF的结合位点,参与调控染色质空间构象。然而,SINEs在神经发育过程中的作用及其调控机制尚不明确。
2024年7月3日,复旦大学脑科学研究院江燕课题组与基础医学院刘赟课题组,在《基因组生物学》(Genome Biology)杂志上发表题为“SETDB1 regulates short interspersed nuclear elements and chromatin loop organization in mouse neural precursor cells(SETDB1调控神经前体细胞中短散在核元件和染色质环构象)”的研究论文,揭示了小鼠神经前体细胞(NPCs)中组蛋白甲基转移酶SETDB1调控SINEs活性和染色质环构象的表观遗传分子机制,及其在NPCs增殖中的作用。
该研究利用ATAC-seq、ChIP-seq、WGBS、Hi-C和RNA-seq等多组学技术,对小鼠NPCs的表观基因组和转录组进行了全方位、多层次的解析。研究结果表明,在NPCs中SETDB1介导的组蛋白H3K9me3修饰,与DNA甲基化发挥协同作用,抑制特定类群SINEs位点的染色质活性。SETDB1敲除后,这群SINEs位点处染色质开放性增加,CTCF与这些位点结合,介导染色质环的重分布。其中,新生成的染色质环参与调控有丝分裂功能通路中的基因表达,从而影响了NPCs的细胞周期和增殖。这一研究揭示了发育脑中SETDB1调控SINEs元件和三维染色质构象的分子机制,并为转座元件参与神经发育提供新的理论依据。
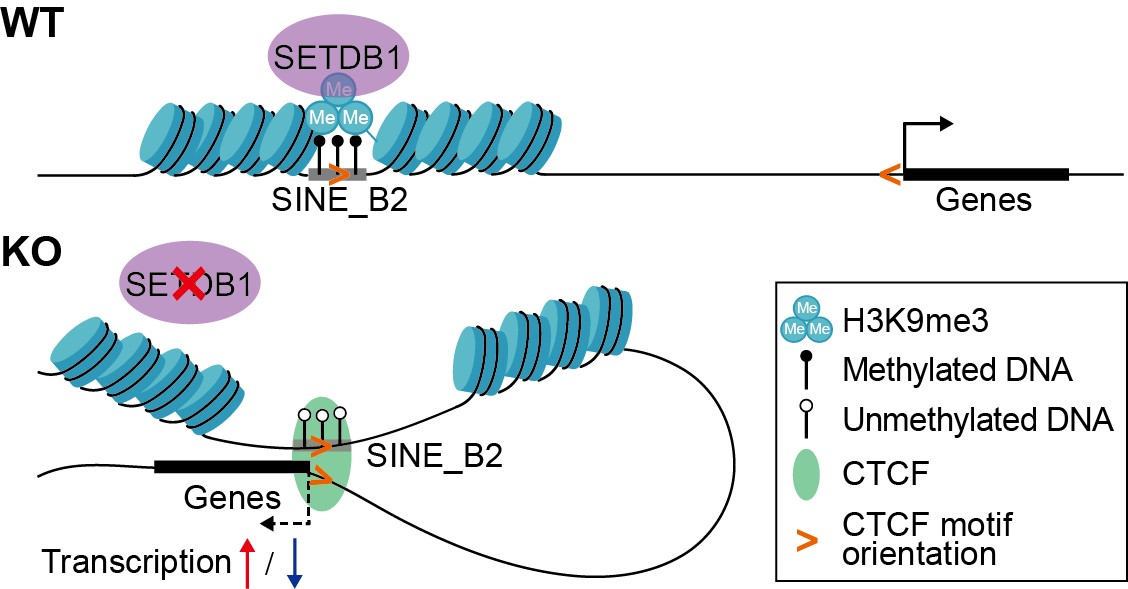
图:SETDB1调控NPCs中SINEs活性和染色质环构象的分子机制
复旦大学脑科学研究院博士生孙戴静、朱月妍和博士后彭文竹为本研究的共同第一作者,江燕研究员和刘赟课题组博士后蒙伟达为共同通讯作者。其他共同作者包括:郑胜辉、翁洁、董书龙、李嘉琪、陈颀、葛传慧、廖立勇、董宇昊和刘赟研究员。该研究得到了科技创新2030重大项目和国家自然科学基金的支持。
论文地址:https://doi.org/10.1186/s13059-024-03327-2
联系方式
地址:上海市东安路131号科研二号楼B栋(200032)
电话:021-54237641
脑科学研究院邀您建言献策,建言献策邮箱:iobs_commun@fudan.edu.cn
友情链接
快速通道
公众号
-
 复旦大学脑科学研究院
复旦大学脑科学研究院
-
 FDU脑海研航
FDU脑海研航
复旦大学脑科学研究院 版权所有








 公共技术平台网上预约
公共技术平台网上预约